当室では、細胞の由来動物種を同定する検査として、DNAバーコーディング検査を実施しております。
この検査はDNA バーコード(特定の遺伝子領域の短い塩基配列)*1 をPCR増幅し、そのPCR産物のDNAシークエンスを行います。シークエンス結果が公開データベース*2 に登録されている各種動物の塩基配列のどれと一致するかを調べることで、動物種を同定することができます。DNAシークエンス解析技術の進歩に伴って、様々な動物種でDNA バーコードのシークエンス解析が実施され、その解析結果が公開データベースに登録されるようになったために動物種同定への応用が可能となった検査方法です。
*1 当室細胞株の由来動物種同定で解析に用いているDNA バーコード
ミトコンドリアのCytochrome c oxidase subunit I (COI) 遺伝子を使用します。
COI遺伝子のみの解析で動物種の同定に至らない場合、その他の遺伝子配列情報が報告されている動物種であれば、Cytochrome b (Cytb) , 16S rRNAなどその他の遺伝子も解析の対象とします。
*2 由来動物種を同定するための公開データベース
Barcode of Life Data Systems(BOLD)
http://www.boldsystems.org/
National Center for Biotechnology Information – BLAST (Basic Local Alignment Search Tool)
https://blast.ncbi.nlm.nih.gov/Blast.cgi
1. 検査の流れ
① DNA抽出 → ② DNAバーコード領域のPCR増幅 → ③ 電気泳動による確認→ ④ 増幅産物の精製 → ⑥ DNAシークエンス → ⑦ 公開データベースによる解析
2. 準備するもの
(1) 検体:細胞から抽出したゲノムDNA溶液
(2) 機器:サーマルサイクラー Applied Biosystems® Veriti® Thermal Cycler 等
(3) PCRプライマー
A)COI遺伝子を増幅するカクテルプライマー Universal primer cocktails ※幅広い動物種に対して増幅が得られます
Mammalian primer mix for routine COI barcoding (Ivanova et al, 2007. Mol Ecol Notes 7:544-548)
※原法ではシークエンス用にM13配列が付与されていますが、当室ではForwardにSP6、ReverseにT7配列を付与して使用しています。
C_VF1LFt1_SP6 Forward primer cocktails (1:1:1:3の比率で混合する)
C_VR1LRt1_T7 Reverse primer cocktails (1:1:1:3の比率で混合する)
B)COI遺伝子を増幅する動物種特異的プライマー ※特定の動物種に対して増幅が得られます
動物種によってはA) のカクテルプライマーでうまく増幅できない あるいは うまくシークエンスが読めない場合があります。
もし検査対象の動物種に特異的なCOI遺伝子プライマー あるいは COI遺伝子の塩基配列情報が公開されているのであれば、特異的プライマーを作製して増幅します。
C) COI遺伝子以外のDNAバーコード増幅用プライマー
COI遺伝子のデータベース登録状況は動物種によって様々です。特に “アジア固有種” や 同じ動物種であっても生息域の異なる “亜種” を同定したい場合、まったく登録がない あるいは 少ない為に、同定に至らない場合があります。
この様なケースでは、対象動物の種・亜種に特異的なその他の遺伝子を用いた解析方法がないか医学文献データベース等で検索し、それらのプライマーを作製して増幅します。
D) シークエンス用プライマー
A) ~ C) の遺伝子増幅用プライマーに、SP6・T7の様な汎用配列を付与している場合は、汎用配列をシークエンス用プライマーとして用います。汎用配列を付与していない場合は、遺伝子増幅用プライマーと同じものをシークエンス用プライマーに用います。
3. 方法
※ ここで紹介するのは「A) COI遺伝子を増幅するカクテルプライマー」を用いる場合です。
その他のプライマーを用いたPCR条件については文献 等をご参照ください。
(1) PCR Reaction Mixture(1反応分)
※シークエンス前にバンドの切り出し精製を必要とする場合は数反応分を用意します。
| 10X Ex Taq Buffer | 5.0 μL |
| dNTP Mixture (2.5 mM each) | 4.0 μL |
| Forward Primer [C_VF1LFt1_SP6] (10 μM) | 0.5 μL |
| Reverse Primer [C_VR1LRt1_T7] (10 μM) | 0.5 μL |
| TaKaRa Ex Taq HS (5 units/μL) | 0.25 μL |
| DDW | 37.75 μL |
|
|
|
| Total | 48.0 μL |
(2) PCRチューブにPCR Reaction Mixture 48 μLを分注し、それぞれに検体の Template DNA 20ng (10ng/μL, 2μL) を加える。
(3) PCRサイクル

(4) 電気泳動による確認 結果例(2% アガロースゲル使用)
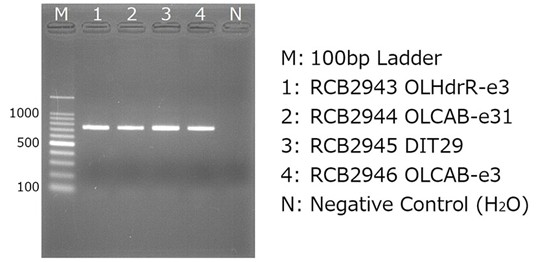
(5) 増幅産物の精製・吸光度測定
市販のPCR精製キット、ゲル切り出し精製キット等を用いて増幅産物を精製します。
また、増幅産物の吸光度を測定しシークエンス解析に必要な濃度に調整します。
QIAquick PCR Purification Kit (50 / 250)(Cat No. 28104 / 28106)
QIAquick Gel Extraction Kit (50 / 250)(Cat No. 28704 / 28706)等
(6) サンガー法を用いたキャピラリーシーケンサーによるDNA配列解析を行います。
配列データと波形データが得られたら、“読み始め” と “読み終わり” の読み取り不良部分をクリーニングしてコンセンサス配列を作成します。

(7) 結果解析
「Barcode of Life Data Systems(BOLD)」に作成したコンセンサス配列を入力して解析します。
解析結果の例:RCB2943 OLHdrR-e3の場合
https://cellbank.brc.riken.jp/cell_bank/CellInfo/?cellNo=RCB2943&lang=Ja
同定された動物種:Oryzias latipes(ミナミメダカ)100%

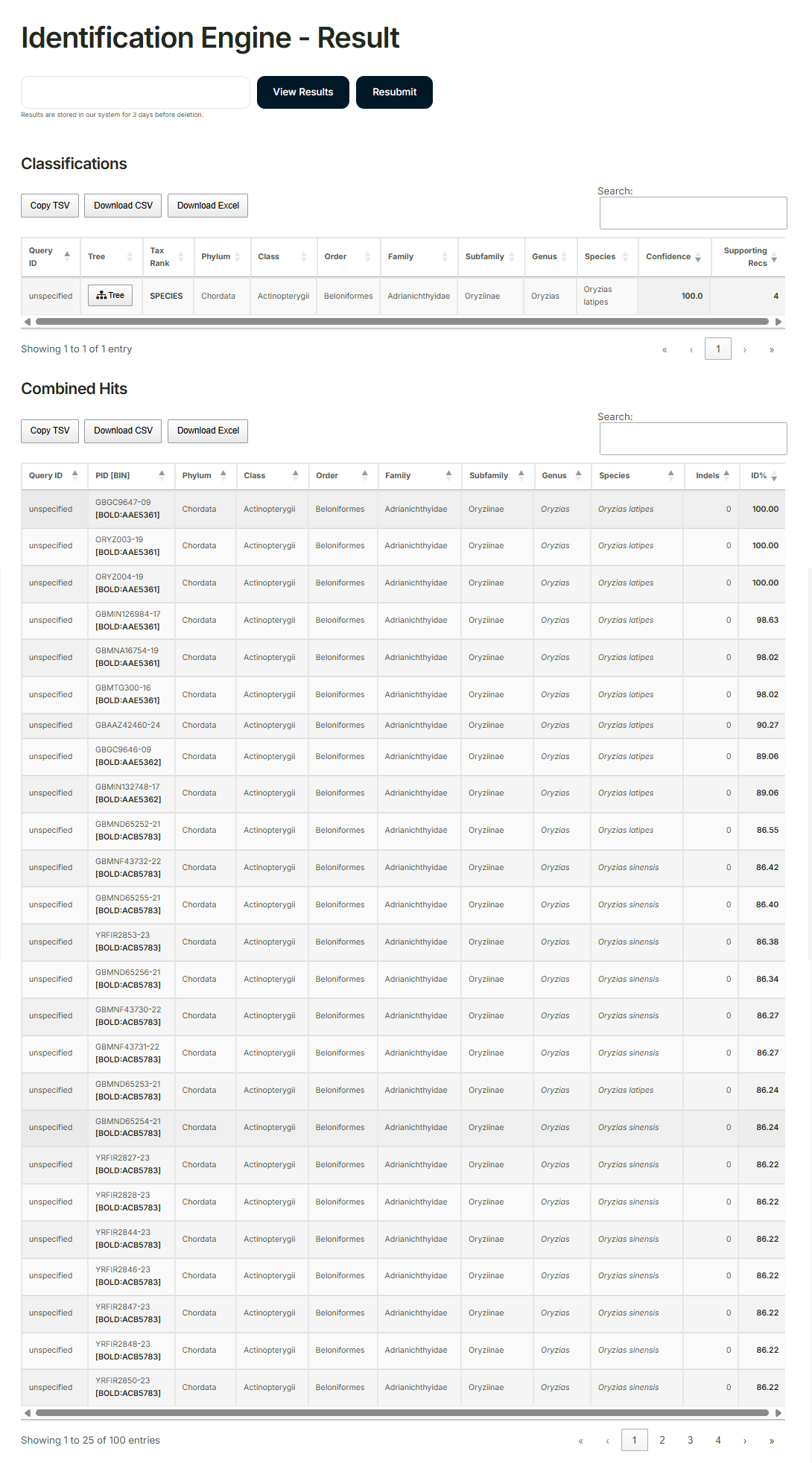
メダカ由来細胞株の解析結果 Oryzias latipes
| ① メダカ(Hd-rR系統) COIシークエンス https://www.ncbi.nlm.nih.gov/nuccore/AB498065 |
⑦ メダカ COI シークエンス https://www.ncbi.nlm.nih.gov/nuccore/NC_004387 |
| ② RCB0187 OLHE-131 Lot.1 :OLHE-131 | ⑧ RCB0184 OLF-136 Lot.1 :OLF-136 |
| ③ RCB2943 OLHdrR-e3 Lot.1 :OLHdrR-e3 | ⑨ RCB0188 OLME-104 Lot.2 :OLME-104 |
| ④ RCB2944 OLCAB-e31 Lot.1 :OLCAB-e31 | ⑩ メダカ(HNI系統) COIシークエンス https://www.ncbi.nlm.nih.gov/nuccore/AB498066 |
| ⑤ RCB2945 DIT29 Lot.1 :DIT29 | ⑪ RCB2942 OLHNI-2 Lot.1 :OLHNI-2 |
| ⑥ RCB2946 OLCAB-e3 Lot.1 :OLCAB-e3 |
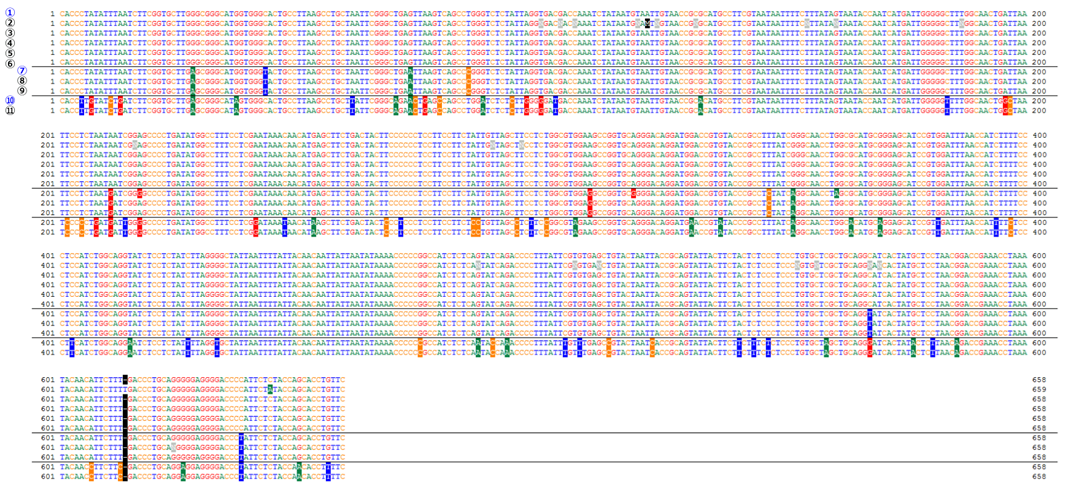
4. 判定
BOLD「Species Level Barcode Records」での解析の結果、95%以上の一致率となり、寄託情報と動物種が一致した場合は合格となります。
「一致率が低い」「データベースの登録情報がない or 少ない」「一致率は高いが亜種の特定まで至らない」
「新たな知見により学名が変更または再分類されている」「学名に対立意見が存在する」など、動物種によって様々なケースがあります。
COI遺伝子だけでは同定できない場合にはその他の遺伝子を用いた解析方法も検討し、総合的な判定を行います。
5. 参考文献
Almeida, J.L., Cole, K.D., and Plant, A.L.
Standards for Cell Line Authentication and Beyond.
PLoS Biol 14: e1002476 (2016).
Hebert PD, Cywinska A, Ball SL, deWaard JR.
Biological identifications through DNA barcodes.
Proc Biol Sci. 2003 Feb 7;270(1512):313-21. doi:10.1098/rspb.2002.2218. PMID: 12614582; PMCID: PMC1691236.





